3D ED / MicroED技術を融合させたスペラファーマの構造解析力
2025.03.03
創薬研究やプロセス開発を強く前に推し進めるための最も重要な課題の一つとして、構造決定があげられる。これをスピーディーかつ確実に実施することで、構造データに基づいた正確な化学的知見により、様々な研究背景を持つ化学者の多角的意見を得ることができる。その結果、次の一手としての明確な研究方針を定めることができ、創薬研究が大きく加速されることになる。スペラファーマでは、基本的な構造解析装置に最新鋭の3D ED / MicroED 1-3) をラインナップに加えることで、より一層の構造解析力向上に努めてきた。本稿では、一般的な構造解析の流れや3D ED / MicroEDを用いた最新の構造解析事例を交えながら我々の取り組みを紹介する。
有機化合物の構造解析の現状
一般的な構造解析の流れを今一度整理するが (Scheme 1)、有機合成化学者であればまず 1H-NMR, 13C-NMR, DEPT135, DEPT 90, 各種2D-NMR, MS, IRなどを測定し、その平面構造 (原子のつながり) を推定していく。簡単な化合物の場合 1H-NMRだけ測定し、所望の構造と推定できればこの時点で合成検討やプロセス検討を前に進めてもおおよそ問題はないだろう。ここで、例えば位置選択的反応や、立体選択的な還元、sp3リッチな多環式化合物の合成等を実施したい場合はあらかじめNOEやNOESYなども合わせて測定しておくべきである。さらに、 1H-NMRシグナルにおけるJ値の解読、カープラス曲線から得られるビシナル水素の二面角を読み解く事によって、最終的に分子の全体像が我々研究者の脳内に鮮明に浮かび上がってくるのである。脳内に形作られた分子模型を参考にすれば、次に実施する試薬がどの部分に反応するのかが予想できる。逆に言えば反応が上手く行かなかった時に、その原因を探るための重要なデータとなるのは言うまでもない。
テルペン系化合物の合成を実施している場合などを例に挙げるが、こうした系統の化合物を取り扱う場合、中心骨格が炭素原子ばかりで形成されている場合が多く、NMRを基盤とする測定のみで化合物の完全な構造決定が出来ない場合も多い。またそういった化合物を標的とする天然物屋さんも構造決定で非常に苦労している。
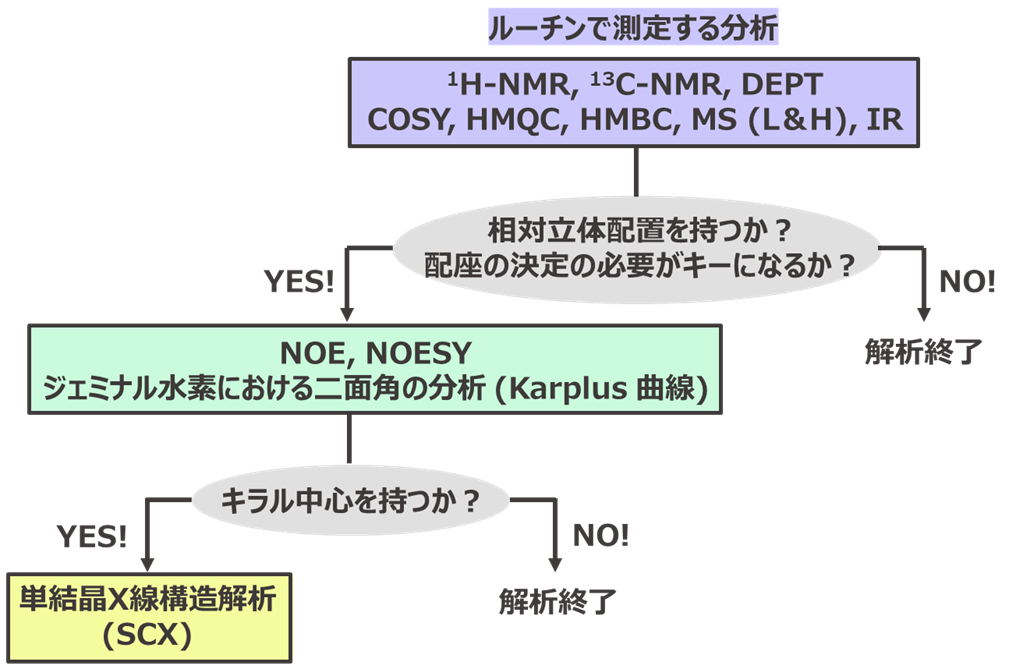
Scheme 1. 一般的な有機化合物の構造解析ワークフロー
結晶構造解析の現状
プロセス開発に従事する研究者であれば、”構造決定” の言葉の範囲がさらに広がってくる。すなわち、原薬 (もしくは中間体も) として得られる結晶に対しても、どのような結晶が出来たのかをしっかりと構造決定する必要がある。通常、結晶には多くのパターンが存在し、1成分からなる結晶、溶媒和結晶、共結晶、塩など様々な様式が知られている (Figure 1)。
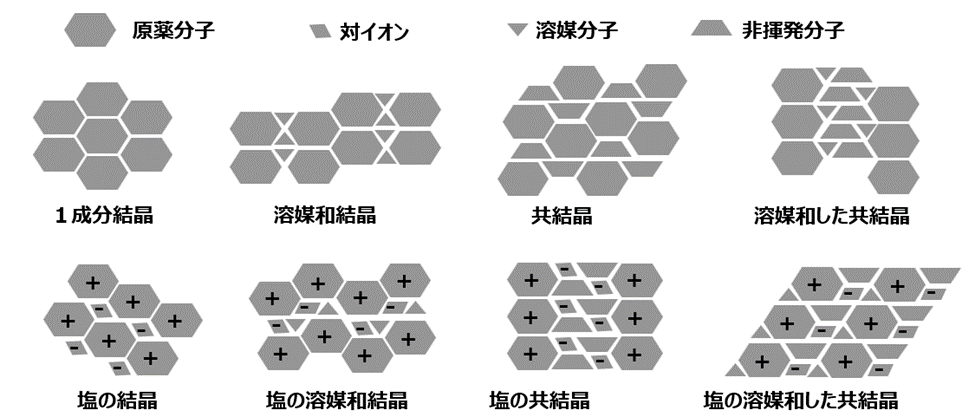
Figure 1. 結晶の分類
どの結晶のパターンにしろ、まずは所望の有効成分 (2成分の場合はコフォーマー、ソルトフォーマー、結晶溶媒、結晶水も含めて) が存在するかどうか確認するため 1H-NMRを測定する。しかし、一度結晶を重溶媒に溶解させてしまうため、それが本当に結晶中に含まれていたかどうか判断することはここでは不可能である。そこで次に、一般的にはPXRD測定が良く利用される (Figure 2)。これにより「結晶である」ことが言え、2成分結晶の場合「ただの混合物ではない」と言うことが証明できる。ただ、多成分結晶の場合、注意すべき点が1つ存在する。それは原薬若しくはコフォーマーとして用いた結晶の多形が運悪く出現し、それが新規の結晶として所望の結晶らしく見えている場合である。我々は日々結晶スクリーニング業務に携わっているが、これは非常に良く出くわす現象である。
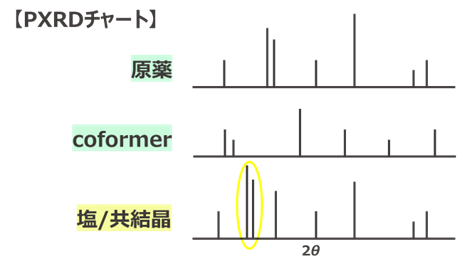
Figure 2. 粉末X線回折分析 (PXRD) を用いた塩/共結晶の推定方法
そこで、最も頼りたくなるのが単結晶X線構造解析である。測定・解析後、単位格子内に所望の分子をORTEP (熱振動楕円体表示) 図として確認することにより一目瞭然で所望の結晶かどうかを判断できる。しかしその測定には、良質かつ比較的大きいサイズの結晶 (100μm程度) を必要とする。創薬研究やプロセス開発を強く前に推し進めるため、研究者は多大な労力をこの単結晶作成に費やしている。見つめる鍋は煮えないにも関わらず、結晶がうまく成長しているかどうか心配になり、晶析用のサンプル管から目を離せなくなる訳である。しかし結晶性物質のうち、単結晶X線構造解析に適したサイズの結晶が得られる割合は3割程度ともいわれており 4)、良質な結晶が得られない場合、残念ながら構造決定を断念せざるを得ない場合も多い。
この状況を打破する目的で、近年単結晶X線技術に関する高度な研究が精力的に展開されている。その結果、回転対陰極型 (ローター型) 高輝度 X線発生装置やシャッターレス半導体検出器開発のブレイクスルーにつながり、現代では数μm~10μm程度の結晶であれば十分構造決定可能な時代に突入している。しかしながら、所謂 “1 μmの壁” が存在し、それ以下の結晶サイズになると、放射光をもってしてもその構造決定は困難である。化合物によっては1 μm以下のパウダー状にしかならない物質も多く、構造決定の完遂を望む場合、誘導化や共結晶化などさらなる労力を費やすことになる。
3D ED / MicroED構造解析について
近年世界的に3D ED / MicroED (マイクロ電子回折) 結晶構造解析が注目を集めている。本測定法は上記 “1 mmの壁” を軽々と超え、有機化合物では100~500 nm程度の大きさがあれば比較的短時間で構造決定可能な革命的な分析手法である。これまで、電子線による回折現象は当然知られていたものの、化合物の電子線損傷が激しく、ディフラクションを得る間に化合物の分解 (結晶性の損失) を伴うことが問題となっていた。しかし近年、高感度の半導体検出器等が開発されたおかげでこれらを回避出来るようになり、短時間の測定でも十分なデータ量のディフラクションを得ることが可能となった。2013年、Gonenらによるタンパク質の3D ED / MicroED構造決定が報告され、大きな注目を集めたことは記憶に新しい 5)。さらに2018年には低分子化合物の構造決定 2,3) が報告され、同年ScienceのBREAKTHROUGH OF THE YEARに選ばれるなど適用事例が増加している。
3D ED / MicroEDの測定方法を以下に示した (Figure 3)。まず銅製の直径 3 mm程度の専用グリッドを準備する。一方測定対象となる結晶をガラス板や乳鉢を用いて細かくすりつぶす。あらかじめ微結晶の場合はその必要はない。次にそのパウダーにグリッドを触れさせ、結晶をマウントする。マウントされたグリッドを適切なホルダーを用いて装置にセットし、その後は電子顕微鏡モードと電子回折モードを切り替えながら最良の1粒を探し、良さそうな結晶 (高角側までしっかりとスポットが出る結晶) が見つかれば本測定に入る。本測定では制限視野電子回折法と1軸回転の手法を組み合わせてデータを蓄積していく。本測定終了後、任意の構造解析ソフトを用いて単結晶X線構造解析と同様に初期構造算出~構造精密化を実施していくイメージである。あらゆる方向からデータの収集を行う単結晶X線構造解析と異なり、3D ED / MicroEDでは原理上1軸回転でしかデータを取得できない。そのため対称性の低い結晶を引き当てた場合は、なかなか初期構造が出なく苦労する場合もある。いずれにせよ、測定自体は比較的短時間で終了する 6)。
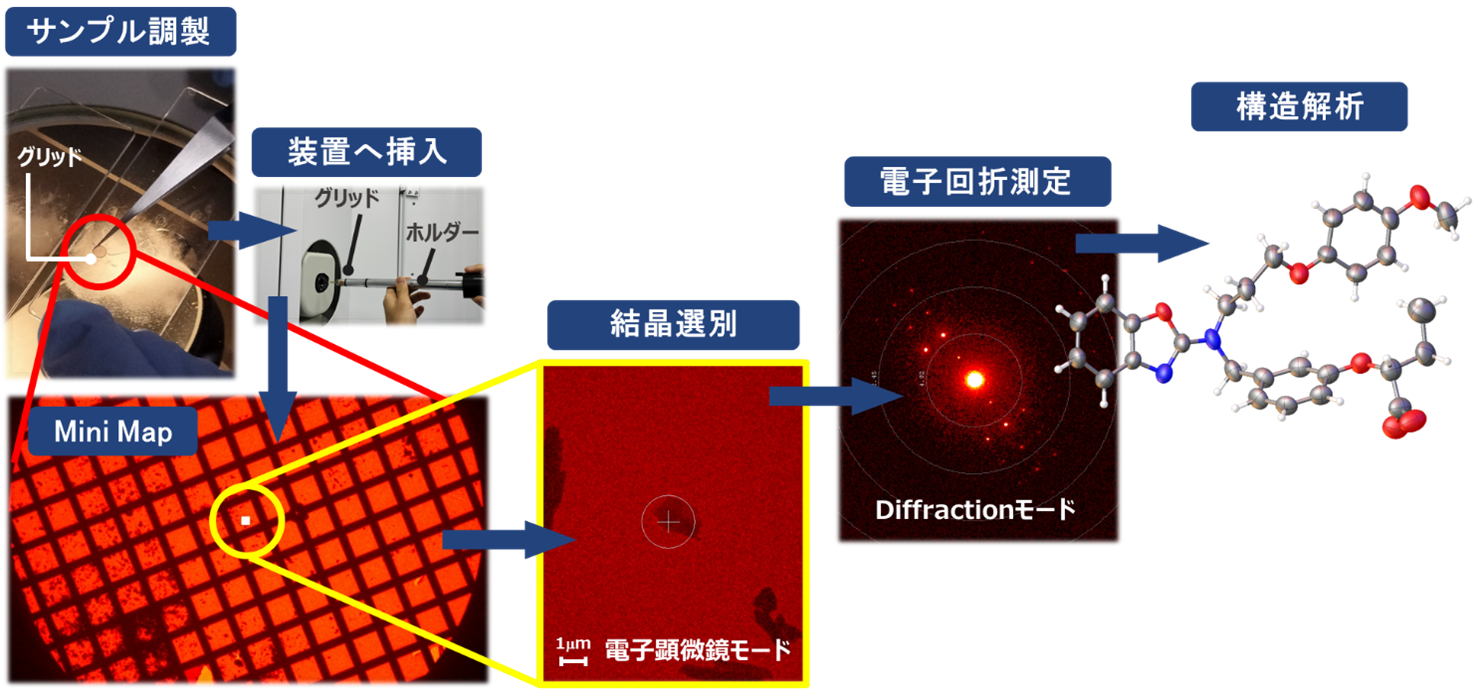
Figure 3. 3D ED / MicroED測定のワークフロー
弊社では、プロセス開発や結晶スクリーニングのさらなるスピードアップ、お客様への高付加価値な結晶学的情報の提供を目的とし、実際にこの3D ED / MicroED装置をリガクより導入した (XtaLAB Synergy-EDⓇ )。以下、単結晶X線構造解析では構造決定困難であった化合物に関して3D ED / MicroED構造解析を進め、多くの実績を得たので紹介する。
塩/共結晶の判別
自らが作成した結晶が塩又は共結晶のどちらかである場合、その最終決定がなかなか上手く行かない場合が多い。”塩” とは酸性物質のプロトンが塩基性物質側に移動し、イオン結合で結晶となった場合を指し、一方プロトン移動を伴わずにそのまま結晶となった場合を “共結晶” と呼んでいる。つまり、塩/共結晶どちらであるのかを判断するためにはプロトンが移動しているか否かを判断すれば良いのである。
ここまでしっかりと構造決定をする意味を良く問われるが、「水素の位置が決まっていない」と言うのはいわば 1H-NMRで水素の位置がよく分からない、と言うのと同じではなかろうか。合成屋さんの中で、水素の数、カーボンの本数が合ってさえいれば良い、と言う研究者も散見されるが、これでは完全な構造決定とは言えない (スピード重視の合成ルート開拓時はその限りではないが)。これと同様に、水素位置の決定による塩/共結晶の推定というものは、化合物の構造決定の完全性という面からも、うやむやにしてはいけない重要な課題となっている。
塩/共結晶の構造決定に関連した各種分析機器についてFigure 4に示した。手軽に利用できる 1H-NMRやPXRDでは、結晶構造の情報が得られないため、基本的には塩/共結晶の判別はできない(Rietveld法を用いれば結晶構造の情報が得られることもあるが)。
そこで、この塩/共結晶の判別にはこれまで3つの手法が特に用いられてきた。
1つ目は単結晶X線構造解析で得られる構造について、カルボン酸部分のC=O, C-Oに関する結合距離を測定する手法である 7)。短い方はカルボニル、長い方が水酸基と判断できるので、そちら側にOHが居るだろうと推定できる。すなわちこの場合共結晶である。逆にもし2つの炭素-酸素間の結合距離が均等な場合、プロトンが電離し平均的な共鳴構造となっていると考えられるため、この場合塩と推定できる。ただし、単結晶X線構造解析に必要な良質な結晶 (結晶性が良く、かつ100 μm程度の比較的大きな結晶) が必要となるため、これが得られなければ測定すら進めることが出来ない。
2つ目は中性子線回折装置の利用である 8)。中性子線は “原子核” そのものとしっかりと相互作用するために、電子をほとんど持たない水素原子であってもその位置をしっかりと決定できる唯一の分析機器である (X線はそもそも電子と相互作用するが、電子を1個しか持たない水素原子に対してはその検出感度が非常に弱い。)。ただし、中性子線回折は放射光設備で実施するため、我々の様な有機化学者はこの装置に容易にアクセスすることが出来ないのが大きな弱点である。
3つめは固体NMRを用いた手法である (NMR crystallography) 9)。プロトン化されうる窒素原子周辺のケミカルシフトを注意深く観察することにより、塩/共結晶の推定が可能とされている。ただこの装置も気軽に使用できるものではない。
以上の様に、従来の分析手法だけでは、塩/共結晶の構造決定は容易にはできない。そこで今回、3D ED / MicroEDによる水素位置決定に基づいた判別の可能性を検討した 10)。
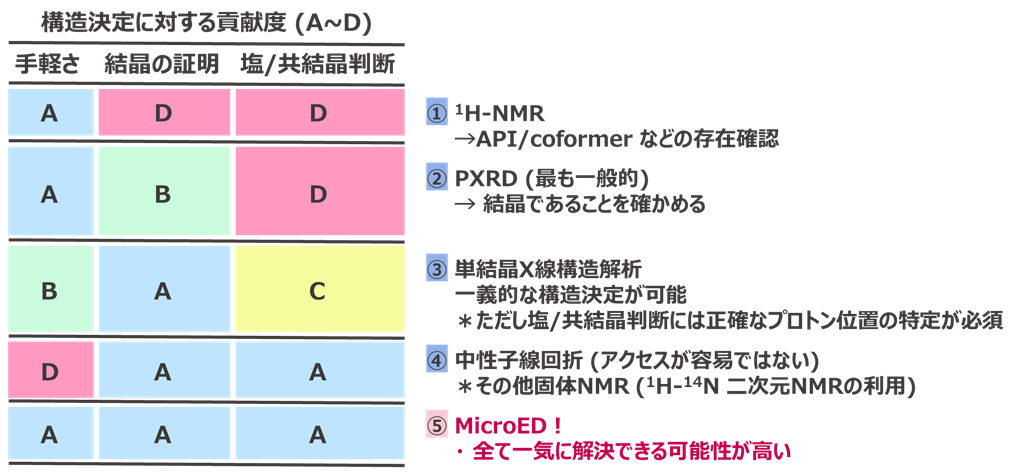
Figure 4. 構造決定に対する各種分析機器の特徴と塩/共結晶の判別
エンシトレビルフマル酸共結晶は、塩野義製薬株式会社によってごく最近開発された新型コロナ感染症に対する治療薬である(Figure 5) 11)。本化合物は塩野義製薬株式会社により、単結晶X線構造解析の手法 (フマル酸のカルボン酸部分の結合距離) を用いて共結晶であると言うことが証明されている 12)。
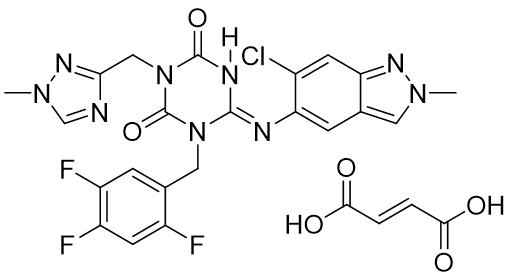
Figure 5. エンシトレビルフマル酸共結晶 (ゾコーバⓇ )
本化合物をモチーフとし、3D ED / MicroEDによる水素位置決定に基づいた共結晶の証明が可能かを検討した。その結果、以下の様にQピーク (電子密度) がカルボン酸周辺にしっかりと観測された。このピークは水素原子として見做せるため、その周辺の結合距離を測定した。その結果、一方の部分では、O38-Q1; 1.044(4) Å vs N9-Q1; 1.558(4) Åであり、もう一方の部分では、O41-Q2; 1.045(6) Å vs N23-Q2; 1.727(4) Åであった。以上の結果から、カルボン酸は電離せずプロトンは窒素原子にプロトンが移動していない、すなわち共結晶であることが分かった。d-Fourier mapによる電子密度表示も、今回の結果が妥当である事を鮮明に表している。エンシトレビルフマル酸共結晶の構造決定は3D ED / MicroEDが水素位置の決定に関しても有効性が高いことを示す好例であろう (Figure 6)。
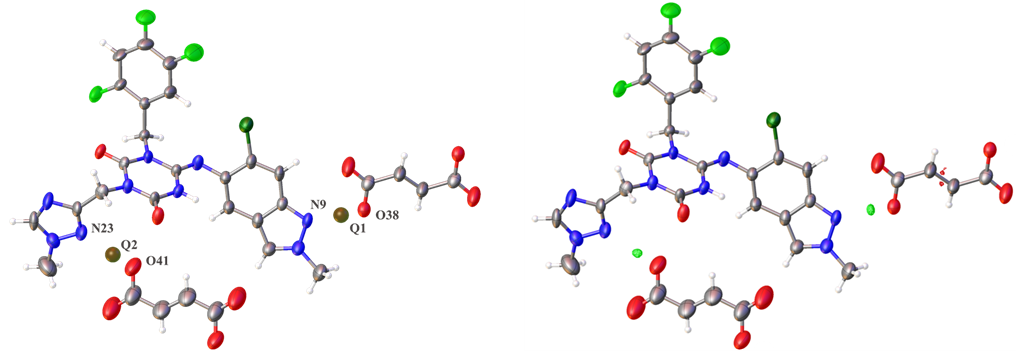
Figure 6. 3D ED / MicroEDによるエンシトレビルフマル酸共結晶の水素位置の特定
(ORTEP図:左 Qピーク表示、 右 d-Fourier map)
結晶多形判別
カソピタントメシル酸塩 (Figure 7) はグラクソ・スミスクライン株式会社によって開発中のNK-1受容体アンタゴニストである 13)。本化合物の結晶多形Form 1, 3は反射型PXRD, 熱分析, IR, ラマンなどの分析法では酷似したチャートを与えるため、これらの分析で両者を見分けることは極めて困難である (Figure 8)。一方、固体NMR及び透過型PXRDのチャートでのみ2つの多形をなんとか区別することができ、これまで単一と考えていた結晶が実は2つの多形の混じり (Form 1と3) であることが明らかとなった。さらに、両者の作り分けは困難であり、純品の結晶多形を得ることは出来ないとされている。
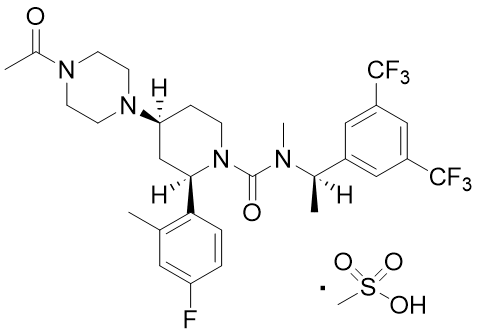
Figure 7. カソピタントメシル酸塩
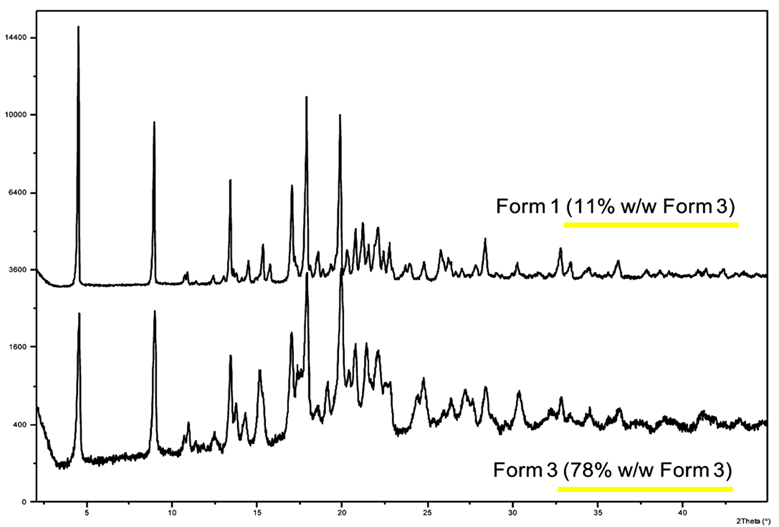
Figure 8. カソピタントメシル酸塩Form 1, 3のPXRDチャート (論文抜粋)
この特異な結晶多形混合物に対して、同一サンプルから一挙に3D ED / MicroEDによるForm1, 3構造決定が可能かどうか検討した。その結果、Form 1 : Form 3 = ca. 9 : 1 の1つのサンプルから、Form 1, 3 それぞれの結晶構造を迅速に導く事に成功した。3D ED / MicroEDでは電子顕微鏡下、制限視野電子回折法により微結晶1粒に電子線を当てながら構造解析出来るため、混合物であっても問題なく分析可能である点が魅力的である。なお、これら分子モデルの重ね合わせの結果、両者の配座が極めて類似していることが分かった (Figure 9)。さらに両者の空間群もP21と同じであったため、物性が酷似していた原因については、おそらく対称性及び配座の類似性に起因すると結論付けることができた。このカソピタントメシル酸塩 “結晶多形混合物” の一挙構造決定は、3D ED / MicroEDの微結晶測定が非常に効力を発揮したことを示す好例である。
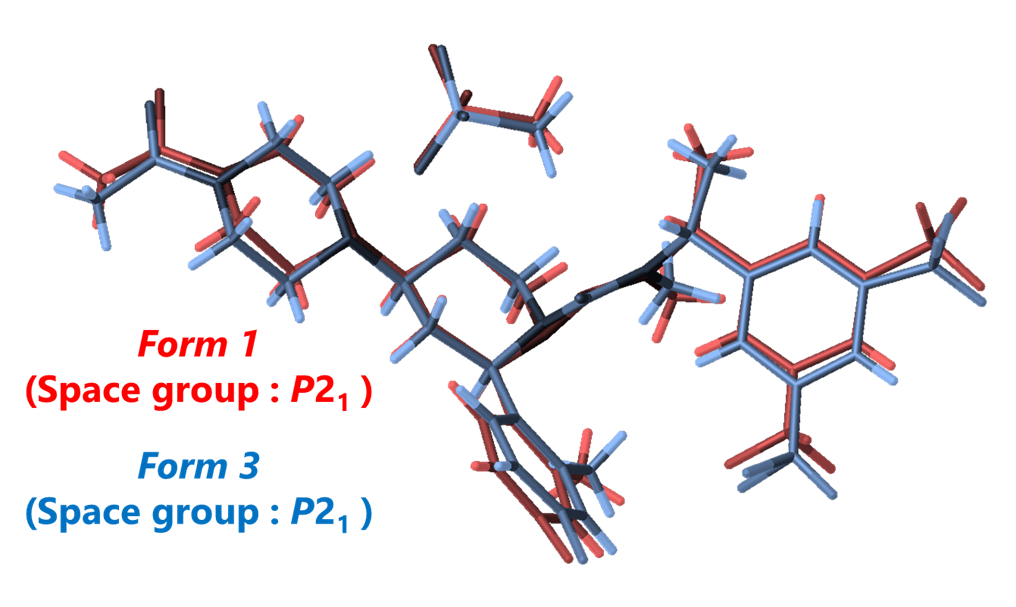
Figure 9. 3D ED / MicroED構造解析で得られたカソピタントメシル酸塩のForm 1と3の立体配座の比較
絶対立体配置の決定
絶対立体配置の決定に関しては、これまでの長い歴史の中で、特に単結晶X線構造解析が主に用いられてきた。単結晶X線構造解析はデータベースや化学組成が不要であり、必要なのは1粒の単結晶だけと言うのが本測定の大きな利点である。単結晶X線構造解析ではFlackパラメータを用いることで絶対立体配置の決定が可能であり、有機合成化学や天然物化学の分野において無くてはならない分析手法である。ただし、前述した通り、所望のサイズの結晶が取得できない場合、この装置の効力を発揮することは出来ない。
3D ED / MicroEDでは単結晶X線構造解析とは異なり、微結晶の測定とKinematicalな解析を実施後、両エナンチオマーの構造モデルを構築させながらDynamicalな精密化を実施することで絶対立体配置の決定が可能となる。基本的には、単結晶X線にしろ、3D ED / MicroEDにしろ、”フリーデル則の破れ” を利用するのだが、3D ED / MicroEDではフリーデル則が破れる原因が多重散乱によるので動力学的回折現象を十分に考慮した精密化 (Dynamical Refinement) を実施する。ここで、Dynamical Refinement後、両エナンチオマーに関してそれぞれR1 (dyn) 値が算出されるが、このR1 (dyn) 値が約1%以上異なっている場合、値の低い方の手系が正しい絶対立体配置であると言われている。
今回、これまで世界的にあまり利用が進んでいなかった”3D ED / MicroED を使用した絶対立体配置の決定”14-16) について、既知天然物 (+)-マンザミンA 17,18) を対象に実証した。
3D ED / MicroED測定および構造解析の結果、(-)-マンザミンAモデルとのR1 (dyn) 値の差は3.82%であり、既知の(+)-マンザミンAであることを確かめることが出来た (Figure 10) [(+)-体; R1 (dyn) 14.01%、(-)-体; R1 (dyn) 17.83%]。このように、複雑な天然物においてもしっかりと絶対立体配置の決定が可能であることを検証できた。
3D ED / MicroEDは、苦労して結晶化検討をしなくとも、エバポレーターで溶媒を留去しただけの結晶性微粉末からでも容易に絶対立体配置の決定が出来るため、各分野の研究者にとって非常に便利なツールとなっていくことは間違いないであろう。
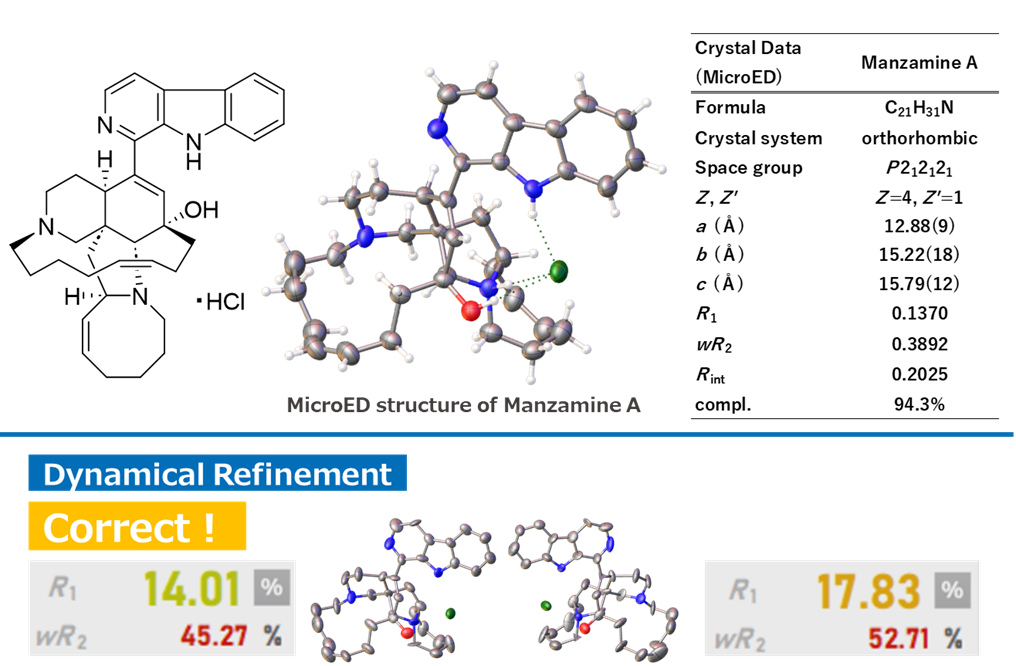
Figure 10. 3D ED/MicroEDによる(+)-マンザミンAの絶対立体配置の決定
おわりに
今回、有機化合物や結晶における構造決定の流れから、様々な分析装置が不得意とする部分についてまで記載した。その上で、各種分析機器の苦手とするパートを上手く補完しつつ、スマートな構造決定の結果を提供してくれる3D ED / MicroEDのパワーをいくつかの例を交えて述べた。3D ED / MicroEDを日常の分析ツールとすることで、弊社でのプロセス合成検討、並びに結晶スクリーニングは以前に比べてはるかにそのクオリティ、スピード感が高まっている。いつの日か3D ED / MicroEDが世界中に普及し、3D ED /MicroED技術に立脚した創薬の時代が訪れることは間違いないが、やはりNMR等を基盤とする分析をしっかりと実施したうえで、そこに3D ED / MicroED技術を融合させることこそが、最も合理的な構造決定のあり方であると筆者は信じている。弊社では引き続き合成技術力の向上、新規合成技術の創出、結晶スクリーニングの高効率化をはじめ、難結晶性化合物の結晶化法開発等、高難度の課題に対しても、お客様により良いソリューションを提供できるよう、改善を続けていく。(中井啓陽、大阪研究センター 製薬研究所)
参考文献
1) Gemmi, M.; Lanza, A. E., 3D electron diffraction techniques., Acta Cryst., 2019, B75, 495-504.
2) Gruene, T.; Wennmacher, J. T. C.; Zaubitzer, C.; Holstein, J. J.; Heidler, J.; Fecteau-Lefebvre, A.; De Carlo, S.; Müller, E.; Goldie, K. N.; Regeni, I.; Li, T.; Santiso-Quinones, G.; Steinfeld, G.; Handschin, S.; van Genderen, E.; van Bokhoven, J. A.; Clever, G. H.; Pantelic, R., Rapid Structure Determination of Microcrystalline Molecular Compounds Using Electron Diffraction., Chem. Int. Ed., 2018, 57, 16313-16317.
3) Jones, C. G.; Martynowycz, M. W.; Hattne, J.; Fulton, T. J.; Stoltz, B. M.; Rodriguez, J. A.; Nelson, H. M.; Gonen, T., The CryoEM Method MicroED as a Powerful Tool for Small Molecule Structure Determination., ACS Cent. Sci., 2018, 4, 1587-1592.
4) C&EN, 2022, OCTOBER, 17th.
5) Shi D., Nannenga B. L., Iadanza M. G., Gonen T., eLife, 2, (2013).
6) Ito, S.; White, F. J., Okunishi, E., Aoyama, Y., Yamano, A., Sato, H., Ferrara, J. D., Jasnowskie, M., Structure determination of small molecule compounds by an electron diffractometer for 3D ED/MicroED., CrystEngComm., 2021, 23, 8622-8630.
7) Aakeröy, C. B.; Fasulo, M. E.; Desper, J., Cocrystal or Salt: Does It Really Matter? Pharmaceutics, 2007, 4, 317-322.
8) Wilson, C. C.; Single crystal neutron diffraction from molecular materials, World Scientific, Singapore, 2000.
9) Rajput, L.; Banik, M.; Yarava, J. R.; Joseph, S.; Pandcy, M. K.; Nishiyama, Y.; Desiraju, G. R., Exploring the salt-cocrystal continuum with solid state NMR using natural-abundance samples: implications for crystal engineering., IUCrJ, 2017, 4, 466-475.
10) Nakai, K.; Miki, K.; Kikuchi, T.; Yamano, M., Detection of Hydrogen Atoms Using Only 3D ED/MicroED and Contribution to Structure Determining Salts or Cocrystals., Growth Des. 2025, 25, 129-135.
11) Unoh, Y.; Uehara, S.; Nakahara, K.; Nobori, H.; Yamatsu, Y.; Yamamoto, S.; Maruyama, Y.; Taoda, Y.; Kasamatsu, K.; Suto, T.; Kouki, K.; Nakahashi, A.; Kawashima, S.; Sanaki, T.; Toba, S.; Uemura, K.; Mizutare, T.; Ando, S.; Sasaki, M.; Orba, Y.; Sawa, F.; Sato, A.; Sato, T.; Kato, T.; Tachibana, Y., Discovery of S-217622, a Noncovalent Oral SARS-CoV-2 3CL Protease Inhibitor Clinical Candidate for Treating COVID-19., Med. Chem. 2022, 65, 9, 6499-6512.
12) Kawajiri, T.; Kijima, A.; Iimuro, A.; Ohashi, E.; Yamakawa, K.; Agura K.; Masuda, K.; Kouki, K.; Kasamatsu, K.; Yanagisawa, S.; Nakashima, S.; Shibahara, S.; Toyota, T.; Higuchi, T.; Suto, T.; Oohara, T.; Maki, T.; Sahara, N.; Fukui, N.; Wakamori, H.; Ikemoto, H.; Murakami, H.; Ando, H.; Hosoya, M.; Sato, M.; Suzuki, Y.; Nakagawa, Y.; Unoh, Y.; Hirano, Y.; Nagasawa, Y.; Goda, S.; Ohara, T.; Tsuritani, T., Development of a Manufacturing Process toward the Convergent Synthesis of the COVID-19 Antiviral Ensitrelvir., ACS Cent. Sci. 2023, 9, 836-843.
13) Cimarosti, Z.; Castagnoli, C.; Rossetti, M.; Scarati, M.; Day, C.; Johnson, B.; Westerduin, P., Development of Drug Substances as Mixture of Polymorphs: Studies to Control Form 3 in Casopitant Mesylate Process. Res. Dev., 2010, 14, 1337-1346.
14) Truong, K.; Ito, S.; Wojciechowski, J. M.; Göb, C. R.; Schürmann, C. J.; Yamano, A.; Campo, M. D.; Okunishi, E.; Aoyama, Y.; Mihira, T.; Hosogi N.; Benet-Buchholz, J.; Escudero-Adán, E. C.; White, F. J.; Ferrara, J. D.; Bücker, R., Making the Most of 3D Electron Diffraction: Best Practices to Handle a New Tool., Symmetry, 2023, 15, 1555.
15) Petricek, L.; Palatinus, J.; Plasil, M.; Dusek, Z., Jana2020 – a new version of the crystallographic computing system Jana., Cryst. Mater., 2023, 238, 271-282.
16) Klar, P. B.; Krysiak, Y.; Xu, H., Steciuk, G.; Cho, J.; Zou, X.; Palatinus, L., Accurate structure models and absolute configuration determination using dynamical effects in continuous-rotation 3D electron diffraction data., Chem., 2023, 15 848-855.
17) Sakai, R.; Higa, T.; Jefford, C. W.; Bernardinelli, G., Manzamine A, a novel antitumor alkaloid from a sponge., Am. Chem. Soc. 1986, 108, 20, 6404-6405.
18) The (+)-manzamine A was gifted by assistant professor Jomori of the University of the Ryukyus as authentic sample.
page top